核酸分子 (DNA Molecular) 的分子動力模擬:
對於研究 DNA 的超螺旋–巨大層級的 DNA 雙股螺旋自行折疊,受到廣泛的注意和興趣。因為超螺旋 DNA 是 DNA 在細胞中具有功能的形式的關鍵:指引和幫助許多基本生物作用的進行,例如複製、重組以及轉錄。這個問題含括在應用上具重要的意義,因為對於 DNA 超螺旋的拓蹼和結構觀點上的瞭解改善,可以影響對於基因治療的載體設計以及影響作為抗癌或抗菌藥物的拓蹼酵素抑制劑。
從實驗裡的短時間尺度上量測 DNA 超螺旋的動力性質是困難的,因此模擬對此能含括過程和機制去研究。由超螺旋性的影響對於序列位並列 (site juxtaposition) 的研究中,使用包含數千個鹼基對的棍狀蠕蟲鏈模型 DNA (DNA 直徑約 2 奈米且拉直後的長度為每 1000 個鹼基對為 340 奈米) ,結合 Brownian 動力理論進行研究。從勢能上可解釋折疊和扭轉變形,對靜電性質的分析,以及流體動力作用力。這樣的結合使得在毫秒時間上模擬的可能,用以解釋影響 DNA 構形柔軟度的關鍵因子。先前對於 site juxtaposition 的預測在考慮時間尺度上不可能量測。這是一個 DNA 分子內部動力造成在空間上將遠距離的線性 DNA 區段帶過來放在一起的過程。
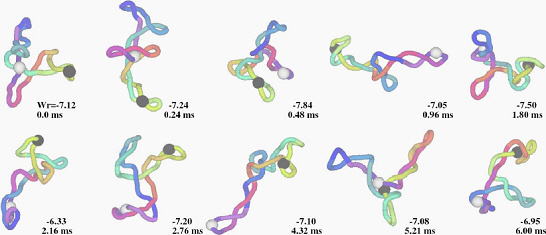
對具有 3000 個鹼基對的 DNA 分子動力軌跡的分析
因此 site juxtaposition 的時間可由超螺旋性,鹽濃度,以及 sites(鹼基位) 間分開的距離的函數來決定。這些量測對於分析特定位置的重組以提供短時間之位置接合的瞭解,且動力過程的機制也可被推斷。